- Intégrer une copie d'écran des groupes emboîtés dans votre compte rendu en cliquant le bouton "Copier / Copier le diagramme (boîtes)".
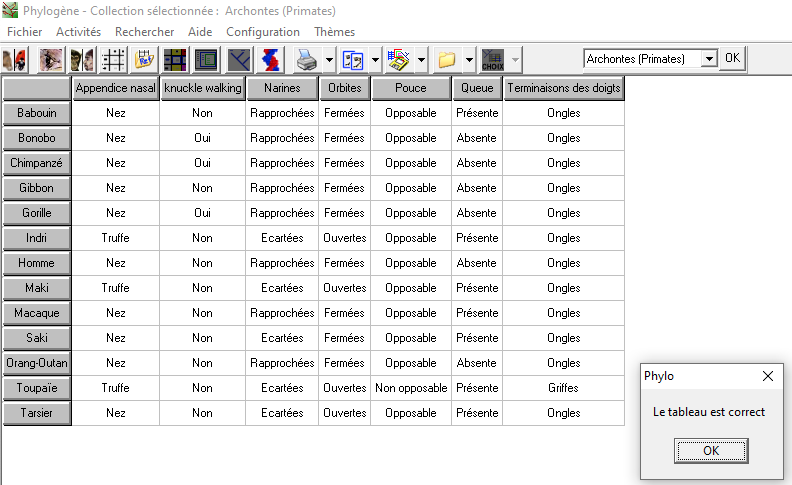
La matrice (ou tableau) de caractères permet de noter, pour chaque espèce choisie, les caractères morpho-anatomiques présents.
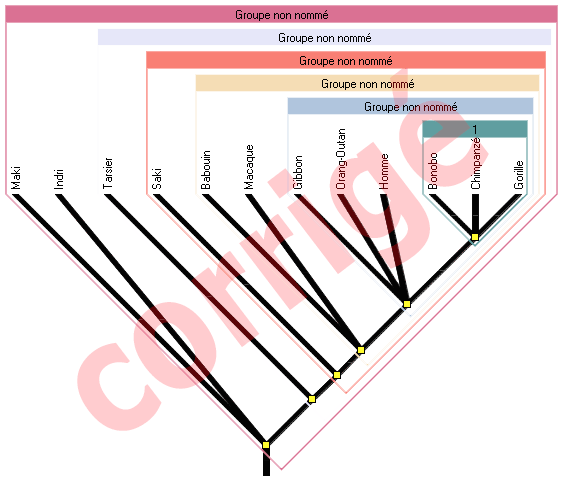
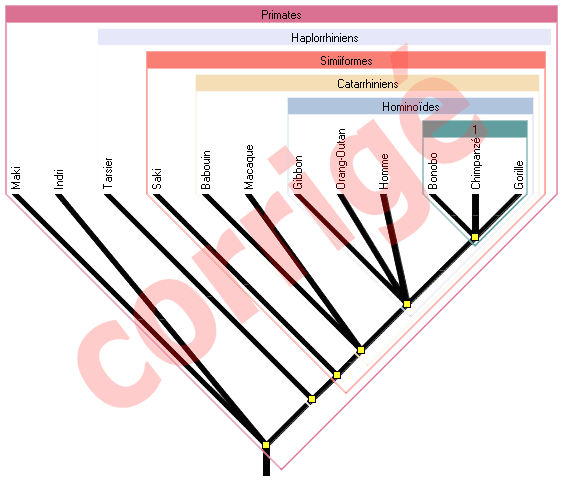
Construction des groupes emboîtés
Le logiciel regroupe les espèces qui partagent les mêmes caractères :
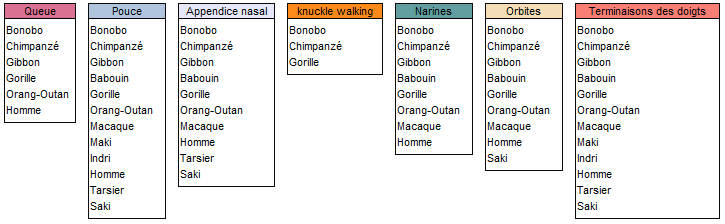
Attention ! Le logiciel indique "queue" pour "absence de queue", "pouce" pour "pouce opposable", appendice nasal" pour "nez", narines" pour "narines rapprochées", "orbites" pour "orbites fermées" et "terminaisons des doigts" pour "ongles"…
Certaines espèces apparaissent dans plusieurs boîtes. Après emboîtement correct :
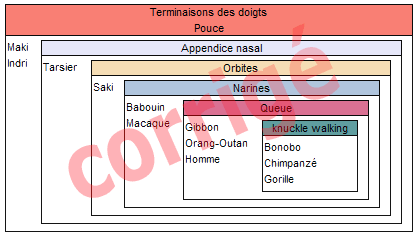
- Dans quelle(s) boîte(s) se trouve l'espèce humaine ? Qu'est-ce-que cela signifie sur notre espèce ?
L'humain se trouve :
- dans la boîte "Hominoïdes" car comme le Gibbon et l'Orang-Outan d'une part, le Bonobo, le Chimpanzé et le Gorille d'autre part, nous n'avons pas de queue (contrairement aux autres animaux pris en compte) ;
- dans la boîte "Catarrhiniens" car comme les espèces déjà citées ainsi que le Babouin et le Macaque nous avons un nez et non un museau (contrairement aux autres animaux pris en compte) ;
- dans la boîte "Simiiformes" car comme les espèces déjà citées ainsi que le Saki nos orbites sont fermées et non ouvertes (contrairement aux autres animaux pris en compte) ;
- dans la boîte "Haplorrhiniens" car comme les espèces déjà citées ainsi que le Tarsier nos narines sont rapprochées et non écartées (contrairement aux autres animaux pris en compte) ;
- dans la boîte "Primates" car comme les espèces déjà citées ainsi que le Maki et l'Indri nos pouces sont opposables aux autres doigts, doigts qui sont prolongés par des ongles et non des griffes (contrairement au Toupaï).
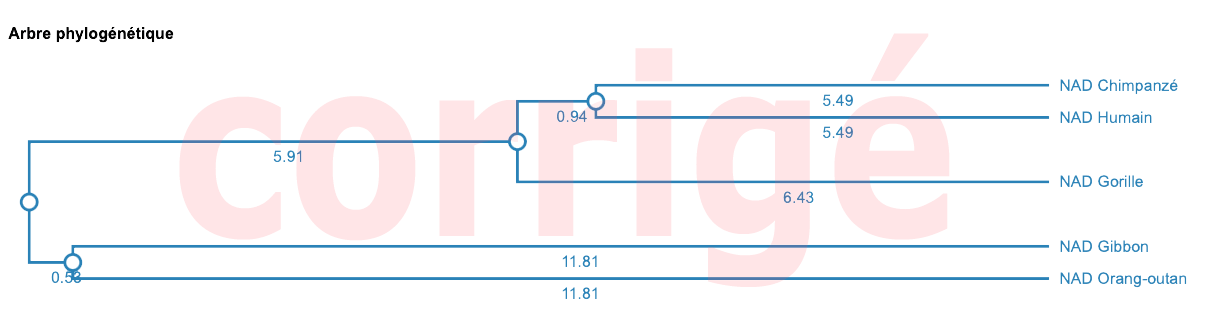
On remarque qu'au sein des Hominoïdes, le Bonobo, le Gorille et le Chimpanzé se trouvent dans un groupe à part qui n'inclut pas l'humain, celui des animaux marchant en "knuckling walking". Est-ce à dire que ces trois animaux sont plus proches entre-eux que de notre espèce ?
- Est-ce en conformité avec les résultats précédents (données morpho-anatomiques) ?
D'après les données morpho-anatomiques, le Bonobo, le Chimpanzé et le Gorille sont plus proches entre-eux que de l'humain. Les deux méthodes ne donnent pas (exactement) les mêmes résultats.
Pour trancher, il faudrait multiplier le nombre de caractères (anatomiques ou moléculaires) pris en compte et les hierarchiser.